感染組織的宏基因組測序,您選對抽提方法了嗎?
本文轉自歐易生物
前言
16S核糖體(ti) RNA基因(rRNA)的擴增測序及分析是評價(jia) 臨(lin) 床標本細菌群落多樣性常用的方法。該方法利用與(yu) 16S r高度保守區域結合的引物,通過PCR擴增16S rRNA基因,對擴增得到的產(chan) 物進行高通量測序及數據分析獲得樣本中細菌群落的OTU,從(cong) 而判斷樣本中的微生物種類。該方法優(you) 點是簡便快速,缺點是大部分微生物無法鑒定到種的水平。
隨著測序平台發展和測序成本的降低,人們(men) 對微生物組成的研究從(cong) 係統發育研究(16S rRNA)轉向了宏基因組測序。通過覆蓋微生物群落的所有物種的基因組信息,使物種鑒定結果具有更高的特異性和敏感性。宏基因組測序還可以提供酶組成或代謝途徑、細菌功能基因組成以及功能與(yu) 係統發育之間的基因組聯係的遺傳(chuan) 證據。另外,宏基因組學不僅(jin) 可以分析來自細菌源的序列,還可以分析來自真菌、病毒和寄生蟲的序列。基於(yu) 上述優(you) 勢,使得直接分析單個(ge) 樣本中的微生物組數據成為(wei) 研究臨(lin) 床樣本微生物多樣性的常用方法。
然而,在研究人類疾病與(yu) 微生物之間的關(guan) 係時,由於(yu) 宿主的汙染,宏基因組測序在實驗上麵臨(lin) 巨大挑戰。宿主與(yu) 微生物的共提取導致後續測序數據中大部分是宿主的基因組數據,從(cong) 而使微生物組的數據不足,還可能產(chan) 生幹擾下遊分析的非特異性信號。解決(jue) 這個(ge) 問題有兩(liang) 種方法:要麽(me) 需要去除宿主,要麽(me) 需要增加測序深度以獲取足夠的微生物基因組數據進行後續分析。然而增加測序深度可能更昂貴,因此我們(men) 比較了幾種去除宿主的微生物基因組抽提方法。
材料與(yu) 方法
1)材料
糖尿病足感染樣本;
2)方法(試劑盒)
實驗設計及結果展示
1. 實驗設計
糖尿病足感染樣本勻漿混樣,平均分成15份,每份25mg,用5種方法抽提,並對所得進行定量PCR及16S r 擴增測序以檢測物種多樣性。

圖1
2. 實驗結果-質量評價(jia)
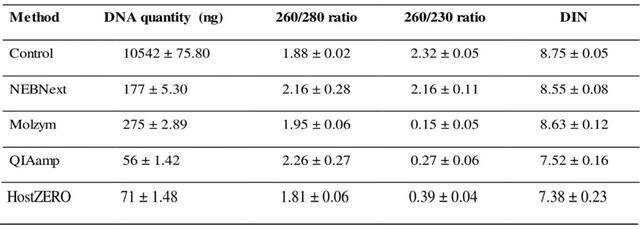
從(cong) 表1可以看出,五種方法抽提的用NanoDrop™2000c(Thermo Scientific)分光光度法測定提取的產(chan) 量和純度約為(wei) 1.8(260/280),基本符合要求。TapeStation 2200顯示完整性(DIN)在7到9之間,這表明沒有碎片化(表1)。而Molzym、QIAamp和HostZERO的260/230比值較低。根據Qiagen應用說明,260/230比率低很可能是由於(yu) 在基於(yu) 柱的試劑盒中裂解緩衝(chong) 液經常使用的胍殘留所導致,但這不影響下遊qPCR檢測。
表1 不同方法抽提的各項指標
3. 實驗結果-成分鑒定
使用細菌16S特異引物(5’-CCTACGGGAGGCAGCAG-3’和5’-ATTACCGCGGCTGCTGG-3’)和人18S特異引物(5’-GGTGGTGCCCTTCCGTCA-3’ 和 5’-CGATGCGGCGGCGTTATT-3’),通過定量PCR檢測樣本中宿主和細菌的占比,結果顯示,NEBNext法的宿主汙染程度高,HostZERO法和QIAamp法的宿主汙染程度低(NEBNext>Molzym>QIAamp>HostZERO)(圖2)對照樣品18S/16S rRNA比值為(wei) 0.865±0.020。而NEBNext法和Molzym法提取的樣本的18S/16S rRNA比值分別為(wei) 0.701±0.022和0.393±0.057,仍然比較高。
相比之下,QIAamp法和HostZERO法的18S/16S rRNA比值較低,分別為(wei) 0.027±0.005和0.015±0.007,分別比對照法低了32倍和57倍。
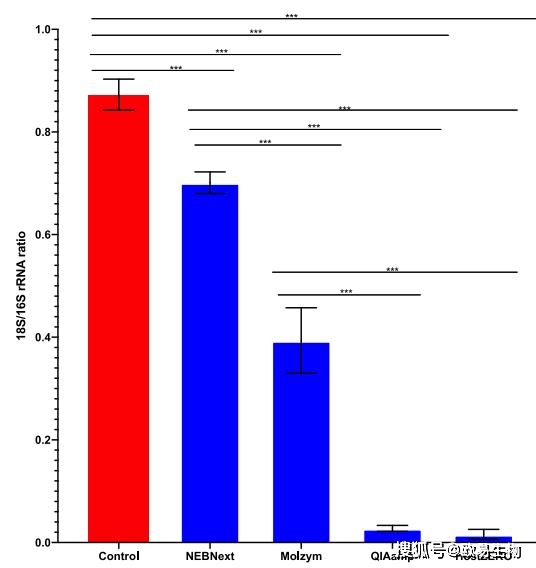
圖2
4. 實驗結果-微生物占比
通過對定量PCR的數據進行拷貝數計算結果表明,對照樣品中細菌含量為(wei) (6.7±0.1%)。在去除宿主後,NEBNext法的細菌占比仍然比較低(8.1±0.2%),Molzym法略高(13.6±1.0%)。在QiaAmp法(71.0±2.7%)和HostZero法(79.9±3.1%)中,細菌含量增加了10倍以上(圖3)。上述結果表明,HostZERO法和Qiamp法能有效地去除宿主汙染,富集微生物。
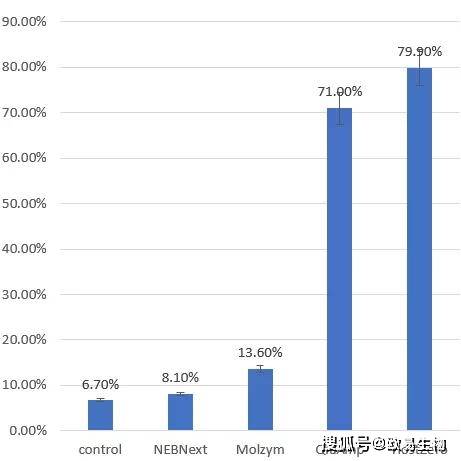
圖3
5. 實驗結果-16S rRNA擴增測序結果
使用16S rRNA V3V4區引物341(5‘-CCTAYGGGRBGCASCAG-3’)和806(3‘-GGACTACNNGGGTATCTAAT-5’)對樣本進行擴增及微生物多樣性檢測,結果表明:糖尿病足感染組織樣本微生物群共包含5個(ge) 門,這些門在該樣本中常見。其中,硬壁門和放線杆菌的成員控製著細菌種群(圖4)。NEBNext與(yu) 對照樣品的細菌物種組成(所有五種報告的菌門的提取)相似,這可能是因為(wei) 這兩(liang) 組樣本的提取方法(Roche)一致,NEBNext另外增加了去宿主的步驟。聚類分析也支持這一點,而HostZERO、Molzym和QIAamp形成了一個(ge) 單獨的聚類(圖4A和圖4B)。在屬一級,鏈球菌主導著糖尿病足感染組織的微生物群,其次是消化鏈球菌(圖4B),與(yu) 已有報道結果一致。總的來說,本研究中使用的所有微生物組富集方法都成功地識別了糖尿病足感染樣本中常見的以及臨(lin) 床上分離出的重要的屬,其豐(feng) 度與(yu) 先前研究中的報告相對相似。
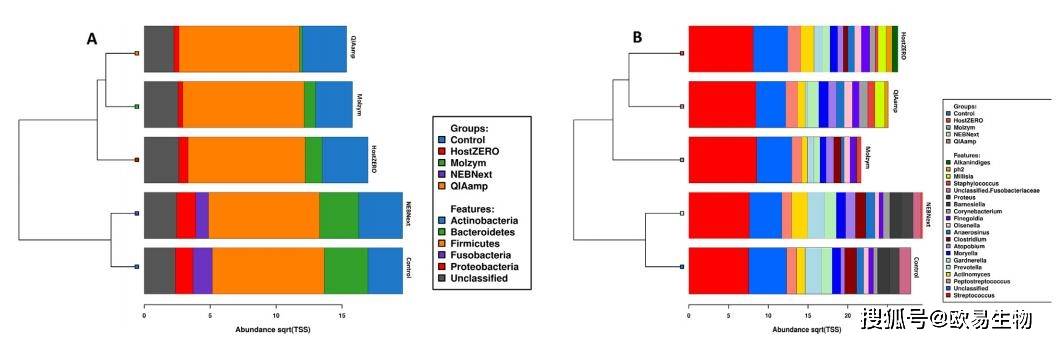
圖4
結論
由以上研究結果可知,在測試的五種抽提方法獲得的質量相近,但是量的差異比較大。不同的去宿主的方法效果不一,以HostZERO的去宿主效果好,QIAamp的效果其次。用這幾種方法抽提的進行16S r擴增對微生物進行檢測,結果一致性較高,但是如果用於(yu) 宏基因組測序,HostZERO和QIAamp抽提方法可能能夠更好的去除宿主基因組的影響。
參考文獻
Host depletion efficiency of microbiome enrichment methods in infected tissue samples, Fatemah Sadeghpour Heravi, Martha Zakrzewski, Karen Vickery, et. Al., 2020, Journal of Microbiological Methods, https://doi.org/10.1016/j.mimet.2020.105856
附表
表2 幾種宿主去除方法的優(you) 劣勢比較

本文轉自歐易生物
電話
QQ掃一掃

